Business Understanding
Jährlich wird in Deutschland bei etwa 293.000 Menschen eine Form von Hautkrebs festgestellt. Bei etwa 35.000 Diagnose handelt es sich um den besonders gefährlichen malignen Melanom, auch als „schwarzer Hautkrebs“ bekannt. Die Prognose der Erkrankung hängt entscheidend vom Zeitpunkt der Diagnose ab. Es gilt: Je früher ein Hautkrebs erkannt wird, desto besser sind die Heilungschancen.
Die Mitarbeit der Betroffenen spielt eine große Rolle. Menschen, die ein höheres Risiko für die Erkrankung tragen, sollten regelmäßig (zum Beispiel einmal im Monat) ihre Haut auf Veränderungen überprüfen und bei Auffälligkeiten schnell einen Hautarzt aufsuchen. Die Unterscheidung zwischen harmlosen Muttermal und Hautkrebs beherrscht nur das Fachpersonal – bisher.
Algorithmen aus dem Bereich Machine Learning und die Verfügbarkeit von Daten über Hautkrebs – in Form von Fotos von klassifizierten Hautkrebsfällen – ermöglichen die Erstellung von Modellen, mit denen auch Laien eine erste Einschätzung einer Hautveränderung bekommen. Einfach, in dem die Stelle auf der Haut fotografiert und dem Modell zur Analyse übermittelt wird. So zumindest die Annahme.
Ziel von diesem Part des „BR Skin Cancer Project“ ist es, solch ein Modell zu generieren. Das „BR Skin Cancer Project“ ist lediglich eine Demonstration der Möglichkeiten. Das Modell wird nicht geeignet sein für echte Bewertungen von Hautveränderungen.
Analytic approach
Die Daten für das Training und Testen des Modells stammen aus dem Kaggle-Datensatz „Skin Cancer MNIST: HAM10000“. Grundlage des Kaggle-Datensatzes sind die Original-Challenge beziehungsweise der Harvard-Datensatz aus der Arbeit „Skin Lesion Analysis Toward Melanoma Detection 2018: A Challenge Hosted by the International Skin Imaging Collaboration (ISIC)“
Das Modell wird mit einem Convolutional-Neural-Network-Algorithmus berechnet, also einem künstlichen neuronalen Netz. CNN ist derzeit so etwas wie der Standard, wenn es um Bilderkennung geht. Mehr Informationen zu CNN gibt es bei Wikipedia hier.
Als Framework wurde Keras ausgewählt, ein High-Level-Framework, das auf Tensorflow aufbaut. Beides sind Open-SourceProgrammbibliotheken, die in der Programmiersprache Python laufen
Data Requirement, collection, understanding
Der Kaggle-Datensatz stellt unterschiedlich aufbereitete Versionen der Hautkrebs-Bilder zur Verfügung. Einmal die Bilder selbst, im jpg-Format. Dazu eine Metadaten-CSV-Datei, die den Zusammenhang der Bilder mit ihrer entsprechenden Klassifizierung auflöst.
Mit diesen Datensätze könnte problemlos gearbeitet werden, das würde jedoch eine umfangreiche Bearbeitung erfordern. Erfreulicherweise liegen die Daten aber auch bereits komplett vorbereitet im CSV-Format vor – und das in zwei unterschiedlichen Auflösungen: Einmal im Format 8 mal 8 Pixel und 28 mal 28 Pixel (jeweils mit und ohne Farbinformationen).
Selbst Letzteres ist eine deutliche geringere Auflösung als die jpg-Bilder, die eine Auflösung von 640 mal 400 Pixel haben. Jedoch: Mit solch einer Auflösung ein neuronales Netz zu berechnen würde die Grenzen dieses Demo-Projekts deutlich sprengen. Dafür müssten entweder kostenpflichtige Rechnerleistung in einem Cloud-Service gemietet oder sehr lange Berechnungszeiten in Kauf genommen werden.
So kommt während der Entwicklung der 28 x 28-Datensatz zum Einsatz.
Der Original-Datensatz enthält die Features „Geschlecht“, „Lokalisierung“, „Alter des Patienten“ sowie „Art der Auswertung“. Bei diesem Modell spielen diese Features keine Rolle. Natürlich kann ein Muttermal bei einer älteren Person anders aussehen als bei einem Jüngeren. Ziel des Modells ist es aber, dass jegliche Muttermale eingeordnet werden, sodass zum Beispiel Alter und Geschlecht dafür nicht relevant sind.
Data preparation
Der Datensatz liegt bereits gut vorbereitet vor. Die Arbeit mit dem Keras-Framework erfordert zuweilen, in Zwischenschritten die Shape umzuwandeln – also die Dimensionen der einzelnen Datensätze.
Für den Einsatz im Keras-Framework wurden die Daten in X- und Y-Datensätzen aufgeteilt. X enthielt alle Bildpunkte, Y das entsprechende Label des Bildes – also, ob das Muttermal unauffällig oder krankhaft war
Wie üblich, wurden die die X-Daten in Trainings- und Testdaten aufgeteilt und normalisiert.
Evaluation
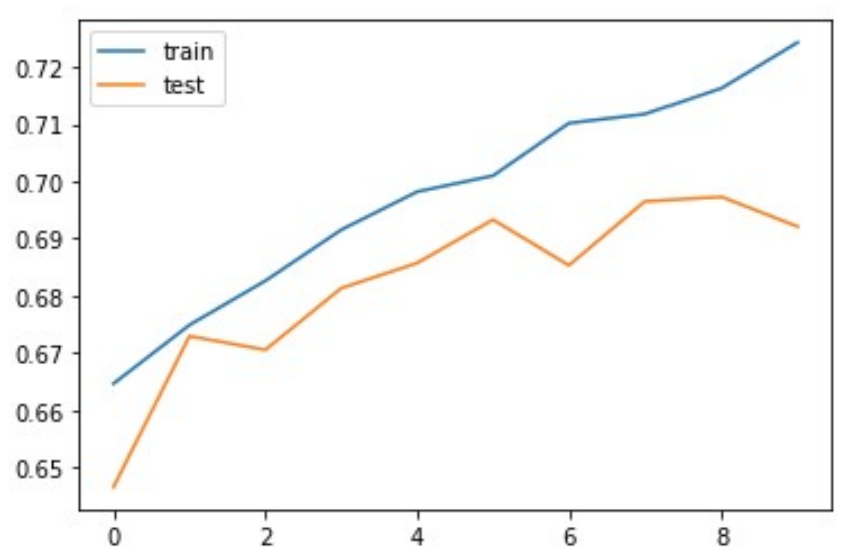
Das CNN wurde mit diversen Hyperparameter-Einstellungen getestet – unter anderem verschiedene Activation-Algorithmen, Layer in unterschiedlicher Anzahl und Feature-Größen, unterschiedlicher Optimizer, etc. Große Variationen kamen dabei nicht heraus. Es pendelte sich ein Accuracy rund um 0.7 bei den Testdatensatz ein.
Allgemein fiel auf, dass das Modell zu einer Überinterpretation der Trainingsdaten neigt – soll heißen, es trat Overfitting auf. Die Accuracy des Trainingsdatensatz lag stets deutlich über den Testdatensatz und stieg kontinuierlich mit der Anzahl der Epochen – ganz im Gegensatz zu der Accuracy bei den Testdaten. Es macht demnach wenig Sinn, mehr als 10 Epochen beim Training des CNN einzusetzen.
Schlussendlich blieb folgendes Setup übrig:
model = Sequential() model.add(Dense(units=14, activation='relu', input_shape=(28,28,1))) model.add(Conv2D(28, (3,3), activation='relu')) model.add(MaxPooling2D(2,2)) model.add(Conv2D(56, (5,5), activation='relu')) model.add(Flatten()) model.add(Dense(units=anz_klassen, activation='softmax')) model.compile(optimizer='SGD', loss='sparse_categorical_crossentropy', metrics=['accuracy']) model.fit(X_train_3D, y_train, epochs=10, batch_size=16, validation_data=(X_test_3D, y_test))
Ein Vergleich der Accuracy von Trainings- und Testdaten als Kurven dargestellt:
Evaluation
Zusammenfassend formuliert: Aus dem Datensatz entstand schnell und unkompliziert ein Modell. CNN ist definitiv der geeignete Algorithmus für solch ein Vorhaben. Dass am Ende nur eine Accuracy von lediglich etwa 0.7 herauskam, ist mit Sicherheit der Auflösung der Daten geschuldet. Die Betrug ja nur 28 mal 28 Pixel, ein doch recht niedriger Wert. Auch die Tendenz zum Overfitting ist wohl Folge der niedrigen Auflösung.
Für die weitere Arbeit an diesem Demo-Projekt soll das hier entstandene Modell ausreichend sein. Nach Fertigstellung der anderen Bausteine wird der letzte Schritt sein, hier an diese Stelle zurückzukommen und die Berechnung des Modells mit voller Auflösung zu wiederholen.
Klick: Komplette Arbeit auf meinem GitHub-Account